Nasce il Covid-19 Data Portal Italy, la piattaforma italiana per la condivisione dei dati genomici sull’epidemia da Coronavirus


Si chiama Covid-19 Data Portal Italy, nasce da una iniziativa del nodo italiano del consorzio europeo Elixir ed è una costola del più ampio portale dell’Unione europea Covid-19 Data Portal. Tutti i dati scientifici raccolti nel continente sul nuovo Coronavirus e la Covid-19 sono destinati a essere catalogati e messi a disposizione dei ricercatori su questo portale. Almeno questo sembra essere il fine ultimo da raggiungere. Si va dalle informazioni emerse nella ricerca scientifica a varie linee guida generali.
«Se stai producendo o lavorando con dati su Covid-19 in Italia e hai domande sulla condivisione e la gestione dei dati, non esitare a metterti in contatto con noi», recita l’appello che si legge sulla home page. Uno dei fronti più importanti della ricerca è sicuramente quello delle varianti Covid e del loro monitoraggio. E questa è la prima cosa che siamo andati a cercare sul portale.
February 2, 2021
Breve guida per chi vuole informarsi sulle varianti Covid
Nella sezione Dati di genomica & trascrittomica troviamo innanzitutto un elenco dei principali database genomici su Sars-CoV-2. La prima risorsa in elenco è ViruSurf, realizzata sotto la guida del professor Alberto Ceri (del Politecnico di Milano). Si trovano dati sui sequenziamenti genomici disponibili, comprese le principali varianti, e su cosa sappiamo delle loro potenziali implicazioni. Il contatore in basso a destra della home indica che al momento abbiamo oltre 200 mila sequenze catalogate. È possibile fare ricerche approfondite in modo da generare tabelle ad hoc. Per gli utenti non è così intuitiva e facile da capire. Ma si tratta di una risorsa importante anche per giornalisti e per chi cerca di fare le pulci alle news sulle varianti.
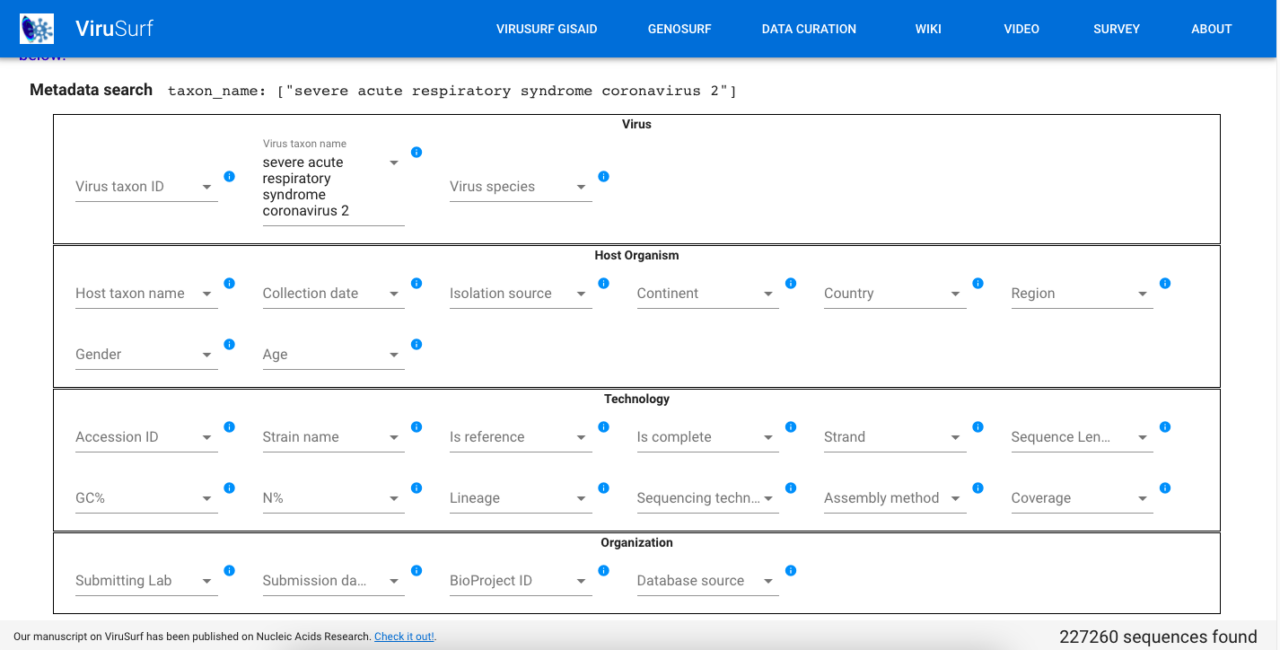
Un altro servizio interessante è Docker4seq. Dà supporto ai ricercatori nell’analisi delle sequenze di RNA, facilitando le cose agli addetti ai lavori meno esperti nel trattare i dati digitalmente.
Sul portale troviamo anche un grafico, aggiornato al 23 gennaio 2021, del sequenziamento genomico nei diversi Paesi, tratto dal database internazionale Gisaid. È interessante notare che i Paesi col maggior numero di sequenze studiate (con grande distacco rispetto agli altri), sono quelli dove sono state trovate le varianti potenzialmente più preoccupanti, a eccezione del Brasile.

Perché è importante monitorare le varianti
La variante 501Y.V1, meglio nota come «variante inglese», presenta a seguito dei sequenziamenti degli ultimi mesi diverse nuove mutazioni. Queste interessano il Rbd (Receptor Binding Domain), la porzione di antigene che lega coi recettori delle nostre cellule. Nel nuovo Coronavirus è la chiave di accesso, soprattutto nei nostri polmoni, ed è nota come glicoproteina Spike (S). Tra le nuove mutazioni più rilevanti A475V, T478I e S494P. Al momento non ci è dato sapere se comportino maggiore infettività.
L’antigene è anche l’elemento che il nostro Sistema immunitario prende di mira, per riconoscere e neutralizzare il virus. Quindi, le mutazioni ci preoccupano soprattutto per quanto riguarda l’efficacia di vaccini e farmaci monoclonali; questi ultimi fatti selezionando appositi anticorpi neutralizzanti. Alcuni studi suggeriscono che questa possibilità ci sia, ma non abbiamo evidenze significative.
Quel che interessa maggiormante è la mutazione E484K, apparsa nelle principali varianti: inglese, sudafricana e una delle due varianti brasiliane. Riportiamo quanto spiega l’esperto di genomica comparata Marco Gerdol in merito:
«Avevo già fatto notare che nel corso degli ultimi due mesi altre mutazioni a carico dell’RBD si fossero già occasionalmente inserite nel makeup genetico del lineage 501Y.V1 (B.1.1.7, VUI202012/01 o “variante inglese” a seconda dei gusti), tra cui A475V e T478I ed S494P – continua Gerdol in un recente post su Facebook – Nessuna di queste tuttavia rappresentava, almeno in linea teorica, un particolare motivo di preoccupazione aggiuntivo rispetto alla maggiore infettività della variante inglese e alla sua rapida crescita in UK, ora piuttosto evidente anche in diversi altri paesi europei».
«L’8 gennaio scorso tuttavia, tra le svariate migliaia di genomi virali sequenziati nel Regno unito, ne era comparso un singolo che recava, oltre alle caratteristiche mutazioni che contraddistinguono B.1.1.7, anche E484K, una mutazione condivisa dagli altri due lineage 501Y emergenti, ovvero 501Y.V2 (la cosiddetta “variante sudafricana”) e 501Y.V3 (la cosiddetta “variante brasiliana”, termine colloquiale che sarebbe meglio abbandonare per motivi che spiegherò sotto)».
Evolution at work Avevo già fatto notare che nel corso degli ultimi due mesi altre mutazioni a carico dell’RBD si…
Posted by Marco Gerdol on Tuesday, February 2, 2021
Queste mutazioni mostrano che le varianti, emerse indipendentemente, presentano qua e là alcuni aspetti sovrapponibili. È evidente una convergenza evolutiva, spiegabile probabilmente con la pressione selettiva di trattamenti con plasma iperimmune in pazienti già immunocompromessi. Quel ch’è certo è che più il virus ha tempo di diffondersi, maggiore sarà la probabilità di avere delle varianti, alcune con mutazioni condivise.
Il fatto che la variante inglese in particolare abbia E484K fa di essa un «doppio mutante», come afferma lo stesso Gerdol. Questo crea un potenziale pericolo. Abbiamo già visto negli studi in vitro che questa doppia mutazione nel RBD della Spike (S), potrebbe aiutare il virus a evadere le difese immunitarie. Senza per questo perdere la capacità di legarsi ai nostri recettori cellulari, in particolare nei polmoni. Se il virus evade le difese immunitarie dovremmo aspettarci dei fenomeni di reinfezione. Effettivamente abbiamo già alcuni indizi in merito, associati alla mutazione E484K. Li abbiamo visti per esempio in Brasile.
La pressione selettiva di trattamenti con plasma di pazienti immunizzati, sembra associata alla presenza combinata di E484K e N501Y. È il caso della variante sudafricana.
«È evidente – spiega Gerdol – che la comparsa di nuove varianti caratterizzate dalla presenza di E484K vada quantomeno monitorata con grande attenzione per le possibili implicazioni epidemiologiche ed immunologiche».
Le ragioni che spingono a un maggiore monitoraggio ci sono e l’esperienza inglese è un esempio importante. Tuttavia, occorre contestualizzare sulla base delle evidenze che abbiamo a disposizione e delle circostanze in cui sono emerse.
«Indipendentemente da queste previsioni, che preferisco lasciare agli aruspici del nuovo millennio – conclude il genetista – Non dobbiamo dimenticarci che le varianti inglese, sudafricana e brasiliana di cui parliamo con grande preoccupazione derivano in origine da un processo di selezione avvenuto in tre soli pazienti fra gli oltre 100 milioni di contagi documentati in tutto il mondo fino ad oggi».
Foto di copertina: La testata del portale italiano del COVID-19 Data Portal.
Leggi anche:
- Anticorpi monoclonali: si aspetta ancora. Dall’Aifa oggi il possibile sì, tra costi e varianti saranno davvero la svolta?
- La risposta di Regeneron ed Eli Lilly alle varianti Covid? Una mappa delle mutazioni che sfuggono agli anticorpi
- Coronavirus, a Varese il primo possibile caso di variante brasiliana in Italia. Si aspetta la conferma delle analisi dell’Iss
- Vaccini in ritardo, caos riaperture: Ricciardi e Galli per la chiusura totale. «Serve un mese di lockdown vero per fermare le varianti»
- Inglese, Sudafricana, Brasiliana (e forse Francese). Ecco le varianti della Covid-19: dove colpiscono, perché sono pericolose, come potremmo combatterle
- Cosa sappiamo davvero della variante «sudafricana»
- Cosa sappiamo delle due varianti «brasiliane» del Coronavirus e perché non dobbiamo (ancora) preoccuparci
