No! In Cina non hanno creato in laboratorio una variante killer della Covid-19


Secondo quanto riportato da diversi media, un team di ricercatori cinesi avrebbe sperimentato una variante killer della Covid-19 mortale al 100% nei topi. La fonte è la prima versione di un preprint del 4 gennaio 2024, nel quale si riporta che il Coronavirus del pangolino GX_P2V «correlato a SARS-CoV-2» avrebbe ucciso tutti i «topi transgenici» da loro infettati. Ma quel che ha reso la ricerca appetibile ai media è probabilmente la parte dell’abstract in cui gli autori suggeriscono «il rischio di propagazione di GX_P2V negli esseri umani». In questo articolo spiegheremo come questo preprint è stato mal interpretato riportando informazioni fuorvianti.
La diffusione fuorviante
Dopo il prevedibile allarme generato, nella seconda versione del preprint questa parte scompare (21 gennaio). Come mai? Un indizio potrebbe essere il fatto che due tabloid in particolare hanno subito dato visibilità con titoli allarmanti alla prima versione: il britannico Daily Mail e l’americano New York Post; generando un effetto domino su diverse altre testate in tutto il mondo, che ne hanno rielaborato i contenuti esaltando le parti più allarmanti.
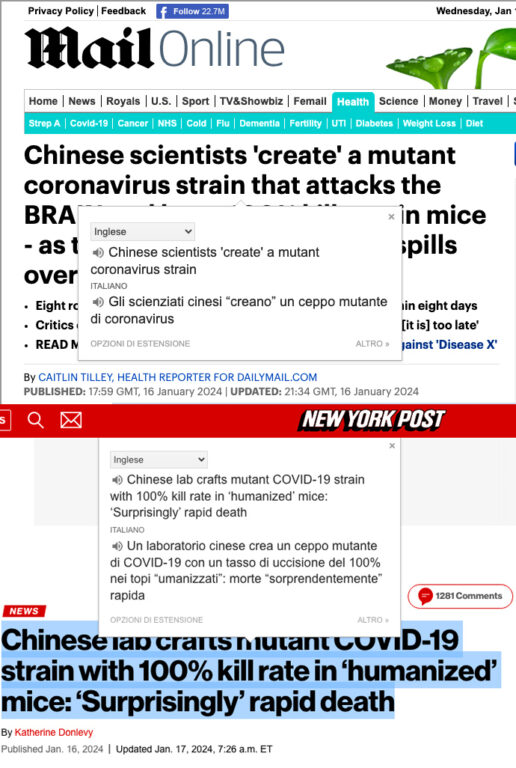
Ecco un esempio di come la prima versione del preprint ha suscitato preoccupazioni tra gli esperti. I media hanno dato grande risalto al commento “a caldo” pubblicato su X dall’infettivologo Francois Balloux, il quale parla di uno «studio terribile»:
Cosa sappiamo del Coronavirus GX_P2V mutato
In realtà – come vedremo -, la ricerca in sé non dimostra alcuna letalità nei topi allo stato naturale, figuriamoci nelle persone. Inoltre si tratta di un Coronavirus scoperto per la prima volta nel 2017. Quindi non è una variante Covid – come si è letto anche in alcune testate nostrane -, e nessuno lo ha creato in laboratorio. Viene descritto anche in uno studio di Nature del 2020. I ricercatori parlano infatti di un «mutante» dello stesso GX_P2V, a cui affiancano la dicitura short_3UTR, che sta a indicare delle particolari porzioni di mRNA mutate nel corredo genetico del virus. Inoltre, non è stato «creato» dai ricercatori – come suggeriscono certi titoli di giornale -, il ceppo infatti si è sviluppato nelle colture cellulari:
«In precedenza abbiamo riportato che l’isolato GX_P2V a passaggio precoce era in realtà un mutante adattato alla coltura cellulare, denominato GX_P2V(short_3UTR) – spiegano gli autori nella prima versione -, che possiede una delezione di 104 nucleotidi al 3′-UTR (6). In questo studio, abbiamo clonato questo mutante, considerando la propensione dei coronavirus a subire una rapida mutazione adattativa nella coltura cellulare, e ne abbiamo valutato la patogenicità nei topi hACE2. Abbiamo scoperto che il clone GX_P2V (short_3UTR) può infettare topi hACE2, con elevate cariche virali rilevate sia nei tessuti polmonari che cerebrali. Questa infezione ha provocato una mortalità del 100% nei topi hACE2. Supponiamo che la causa della morte possa essere collegata al verificarsi di un’infezione cerebrale tardiva».
Da un totale di otto cloni del Coronavirus mutato i ricercatori hanno selezionato casualmente il settimo, denominato «GX_P2V C7». Quindi sono stati usati dei topi modificati geneticamente in modo da esprimere una grande quantità di recettori ACE2 umani (presenti nelle cellule, sono i principali bersagli che permettono al virus di infettarle). Questo è un punto importante. Infatti gli stessi autori citano un precedente studio di Shi Zhengli del gennaio 2023, dove i topi in condizioni normali non venivano uccisi dall’infezione. Ricordiamo che si tratta della scienziata tra le più impegnate nello studio dei BetaCoronavirus dei pipistrelli e dei pangolini, fin da prima della pandemia di Covid-19. Parliamo di quattro topi uccisi dal mutante. Per il controllo sono stati inoculati quattro topi col virus inattivato, mentre altri quattro erano stati «mock-infected».
«Sorprendentemente – continuano i ricercatori -, tutti i topi infettati dal virus vivo sono morti a causa dell’infezione entro 7-8 giorni dall’inoculazione, con un tasso di mortalità del 100% […] Abbiamo rilevato quantità significative di RNA virale nel cervello, nel polmone, nei turbinati, nell’occhio e nella trachea dei topi infetti […], mentre è stata rilevata una quantità bassa o assente di RNA virale in altri organi come cuore, fegato , milza, reni, lingua, stomaco e intestino.».
Differenze tra prima e seconda versione del preprint
Ora vediamo come è stato modificato lo studio. Per poter evidenziare tutte le parti che differiscono tra le due versioni del preprint abbiamo usato Copyleaks, un piattaforma online per l’analisi dei testi basata sull’intelligenza artificiale. Abbiamo evidenziamo in grassetto come è stato aggiornato l’abstract, dove scompare il pericolo di propagazione negli esseri umani e la ricerca passa dall’essere un prezioso modello per comprendere la patogenicità dei virus correlati a SARS-CoV-2 a un «surrogato» per valutare l’efficacia di farmaci e vaccini:
Prima versione dell’abstract (2 gennaio)
«Il coronavirus pangolino correlato alla SARS-CoV-2 GX_P2V (short_3UTR) può causare una mortalità del 100% nei topi transgenici umani ACE2, potenzialmente attribuibile a un’infezione cerebrale in stadio avanzato. Ciò sottolinea il rischio di propagazione di GX_P2V negli esseri umani e fornisce un modello unico per comprendere i meccanismi patogeni dei virus correlati alla SARS-CoV-2».
Seconda versione (21 gennaio)
«Il coronavirus pangolino correlato alla SARS-CoV-2 GX_P2V (short_3UTR) è altamente attenuato, ma può causare mortalità in un modello murino umano transgenico ACE2 appositamente progettato, rendendolo un prezioso modello surrogato per valutare l’efficacia di farmaci e vaccini contro la SARS-CoV -2».
Da un titolo allarmante a uno più asettico
Anche il titolo cambia sensibilmente passando dal preoccupante «Infezione letale di topi umani transgenici ACE2 causata dal Coronavirus del pangolino GX_P2V correlato al SARS-CoV-2 (short_3UTR)» a un più asettico «Un modello murino di infezione e patogenesi nel Coronavirus del pangolino GX_P2V correlato al SARS-CoV-2 (short_3UTR)».
Nella pubblicazione del 2 gennaio troviamo dopo l’abstract una lunga lettera al potenziale editore interessato alla pubblicazione. Un preprint infatti è un testo in attesa di trovare una rivista scientifica, che – previa verifica mediante la peer-review -, ne approvi la pubblicazione. Nel testo del 21 gennaio tale lettera si divide in tre paragrafi: Introduction; Results e Discussion.
Le modifiche nei «Risultati»
La parte della lettera originale che diventa il paragrafo Results nella seconda versione presenta delle modifiche nel seguente passaggio:
«Il mutante GX_P2V(short_3UTR), inizialmente isolato dai primi passaggi del campione GX_P2V (6), e il virus GX_P2V stesso, non sono stati studiati in termini di mutazioni adattative nelle colture cellulari. Per ottenere un clone geneticamente omogeneo per esperimenti su animali, abbiamo clonato il mutante passato attraverso due successivi test sulla placca» (prima versione).
«Per prima cosa abbiamo analizzato le mutazioni adattative del mutante GX_P2V (short_3UTR) nelle colture cellulari mediante clonazione e sequenziamento casuali. Il mutante passato è stato clonato attraverso due successivi saggi sulla placca» (seconda versione).
Le modifiche nella «Discussione»
Nella prima versione, corrispondente alla parte che diventerà il paragrafo Discussion leggiamo i seguenti passaggi:
È importante notare che non abbiamo isolato il ceppo GX_P2V di tipo selvaggio. Lo studio di Zhengli Shi et al ha testato la variante GX_P2V(short_3UTR) che abbiamo segnalato. Tuttavia, i cambiamenti evolutivi adattativi di questa variante durante la loro coltura di laboratorio rimangono poco studiati. […]
A causa della propensione dei coronavirus a subire mutazioni adattative durante il passaggio della coltura, abbiamo clonato e analizzato le mutazioni in GX_P2V (short_3UTR), concentrandoci specificamente sulla patogenicità dei ceppi clonati. Il meccanismo ad alta patogenicità di GX_P2V C7 nei topi hACE2, in assenza del controllo GX_P2V di tipo selvaggio, richiede ulteriori indagini. Rispetto alla sequenza originale di GX_P2V(short_3UTR), GX_P2V C7 presenta due mutazioni di aminoacidi nella proteina spike. Data la stretta relazione tra la virulenza del coronavirus e le mutazioni della proteina spike (7), è possibile che GX_P2V C7 abbia subito una mutazione che potenzia la virulenza. Tuttavia, è importante notare che il nostro modello di topo hACE2 potrebbe essere relativamente unico. L’azienda non ha ancora pubblicato un articolo su questo modello murino hACE2, ma i nostri risultati suggeriscono che hACE2 potrebbe essere altamente espresso nel cervello del topo. […]
In sintesi, il nostro studio fornisce una prospettiva unica sulla patogenicità di GX_P2V e offre un modello alternativo distinto per comprendere i meccanismi patogeni dei coronavirus correlati alla SARS-CoV-2.
Le modifiche della seconda versione nella «Discussione»
Nella seconda versione il paragrafo Discussion presenta invece i seguenti passaggi:
«È molto probabile che l’elevata patogenicità di GX_P2V C7 nei nostri topi hACE2 sia dovuta alla forte espressione di hACE2 nel cervello del topo. In circostanze normali, sia il cervello umano che quello dei topi mostrano una bassa espressione di ACE2 (6, 7). Inoltre, anche se la società non ha ancora pubblicato un documento che dettaglia la costruzione e la caratterizzazione di questo modello murino hACE2, ci viene comunicato che questi topi hACE2 hanno una fisiologia anormale, come indicato da livelli sierici di trigliceridi, colesterolo e lipasi relativamente ridotti, rispetto a quelli di topi C57BL/6J di tipo selvaggio. Pertanto, i risultati delle infezioni dei topi in questo studio non hanno alcuna correlazione con le infezioni umane e non alterano la natura fondamentale di GX_P2V(short_3UTR) in quanto altamente attenuata».
«Attualmente esiste l’urgente necessità di sviluppare vaccini ampiamente protettivi contro il pan-SARS-CoV-2, ma l’emergere della prossima variante SARS-CoV-2 è imprevedibile. Il coronavirus del pangolino GX_P2V(short_3UTR), che condivide un certo grado di omologia con SARS-CoV-2, può essere utile per valutare l’efficacia dei candidati vaccini COVID-19 ad ampio spettro contro varianti future sconosciute. Inoltre, il nostro modello di infezione letale nei topi non presenta risposte infiammatorie evidenti nei principali organi colpiti, polmoni e cervello, fornendo così un modello alternativo per valutare l’efficacia dei farmaci antivirali nell’inibire la replicazione virale in vivo».Insomma, dopo una prima versione in cui si suggeriva un vago pericolo di propagazione negli esseri umani di un mutanti di GX_P2V, nella seconda si chiarisce in maniera più chiara che non esiste una correlazione tra le infezioni dei topi e quelle umane. Il fatto che questo Coronavirus risulti fortemente attenuato viene inoltre precisato con maggiore chiarezza. Non di meno, anche leggendo la prima versione appare chiaro che il preprint ha dei limiti tali da non poter dimostrare alcuna pericolosità in condizioni naturali.
Leggi anche:
- Telepathy: il primo microchip di Neuralink trapiantato in un essere umano. Musk: «Risultati promettenti»
- Usa, uomo sopravvive tre giorni collegato a un fegato di maiale
- Le fantomatiche “rivelazioni del medico ONU” su grafene e luciferasi nei vaccini
- Cosa sappiamo dell’incremento di alcuni tumori nei giovani
- «La Cina ha isolato e mappato il Coronavirus due settimane prima di rivelarlo al mondo»
- Questo manifesto su Telethon e la sperimentazione animale è fuorviante
- Le vecchie e nuove bufale sull’Aids (e chi le ha diffuse)
